Course Project
title: QM/MM calculation of potential-energy curve of target enzyme catalytic reaction and enzyme design adapted to the novel 4+4 cycloaddition reaction
Introduction
在生物界中,存在着大量的酶催化反应。在我们探索各种不同的酶催化反应的过程中,我们更多的聚焦于两大问题:如何提高酶催化反应的效率和如果赋予酶更多的功能。根据最近的科学研究,对于第一个问题:如何提高酶催化反应的效率,现在已经有多种解决的方法,其中最负盛名的便是获得2018年诺贝尔化学奖的由Frances H. Arnold开发的蛋白质定向进化方法。然而,如何赋予酶新的功能,或者说如何基于蛋白质设计来赋予酶催化反应不同的反应途径这个问题一直没有得到一个好的答案。如何寻找到一个通用有效的方法来改进酶结构,使得酶能够发生不同的酶催化反应成为了我们主要研究的目的。
根据现有的研究表明,metalloesterase MID1sc10拥有能够催化3-vinyl indole与azachalcone的Diels-Alder[4+2]环加成反应。但这个过程在酶中是分步进行的。具体的反应式如下:

为了探究这个反应在酶中与在无酶状态下反应过程的不同,并给予反应机理进行酶设计使其能够进行全新的反应,我们利用Gaussian09量子化学工具对反应过程与酶结构进行了分析。
Potential Energy Curve Calculation
MD Simulation
首先我们对蛋白质结构进行了模拟,初始结构为包含中间体HAD小分子的晶体结构。从X-ray晶体结构出发进行了100ns的模拟,使用的力场为AMBER套件中的ff14SB。模拟结果利用CPPTRAJ进行分析,导出轨迹文件,并做构象聚类分析得到占比前十的构象。
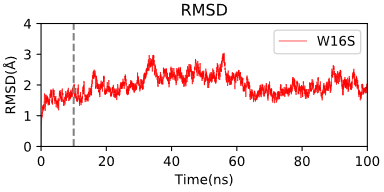

Figure 1 分子动力学模拟RMSD结果与聚类分析结果中前五的构象
在后续的分析中,我们发现在MD的结果有以下特点:
-
聚类的构象中大部分小分子空间结构已经失去了与Zn的结合,而根据先前的调查结果表明,酶中的锌离子是分步反应进行的关键,所以大部分的聚类结果都不能够发生反应。
-
反应过程中蛋白质的构象变化并不大,很快的时间后便达到了较为稳定的状态
最后,考虑到在模拟开始不久的几帧中小分子与锌结合的较为合理,并且已经达到较为有活性的构象,我们选取了模拟开始的前3帧作为QM模拟的起始文件。
QM/MM simulation
在这个过程中我们使用的方法为ONIOM方法。在第一步优化时我们尝试了不同的蛋白质结构,发现在我们集中选取的MD前几帧结构对产物结果影响较小,未发现明显的产物变化。
从MD的结果出发,我们进行了opt优化,得到了对应六元环Hetero-Diels-Alder反应的产物。根据我们的推断,我们认为MD得到的结果中的小分子的构象应当为反应过程中中间体的构象,但此时优化中间体却难以成功。
我们得到的势能面结果
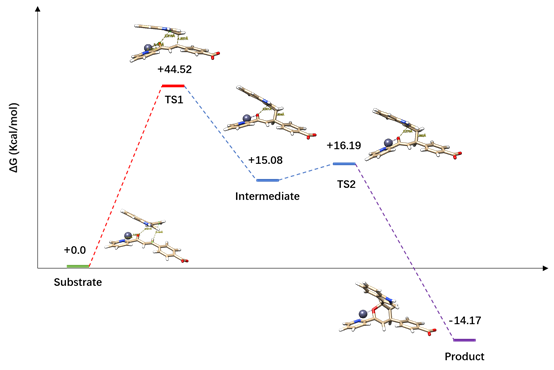
Protein Design
备选反应选择
在之前的研究中发现,同样的反应物也能够发生另一个较为特别的反应:4+4环加成反应,具体反应式如下:
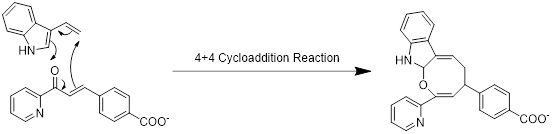
比较4+4环加成反应与之前在酶中发生的4+2HDA反应理论上的机理,我们发现两者的机理存在很大的相似性,而且Zn金属离子在其中发生的作用是相近的:
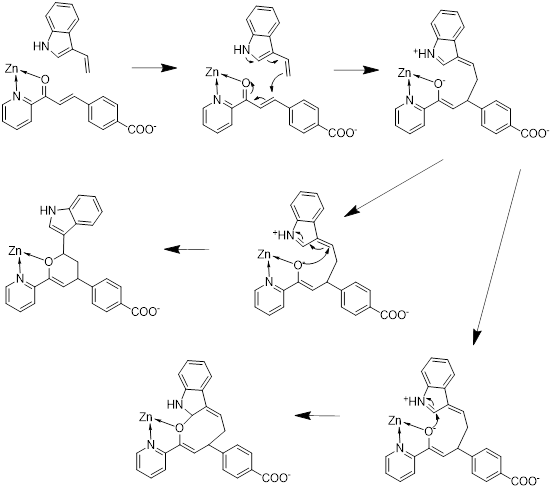
两者的机理中,Zn均起到了稳定中间体中电荷的作用,使得反应能够分步发生,这样也使得反应的过程不再是协同过程,而是两个分步进行的Micheal加成反应。
催化中心周围空间结构分析
比较两个反应发生差异的中间体结构,我们能够发现,4+4反应中,上方吲哚分子的摆放位置有很大的变化,这应该是4+4反应能否发生的关键。而在酶中,蛋白质的氨基酸侧链一定程度上限制了吲哚分子的旋转空间:如下为反应中间体时酶与受体分子的作用关系网络:
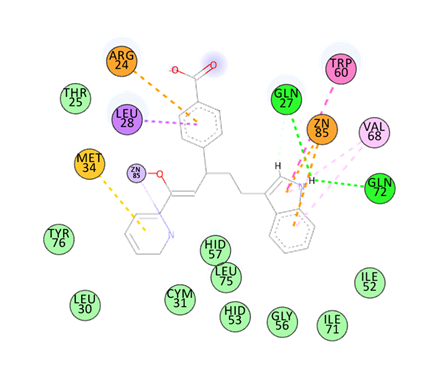
在这个网络中,吲哚分子主要受到临近残基57HIS组氨酸的限制,于是我们决定突变掉这个氨基酸,使用Swiss-Model对蛋白质进行同源建模,然后调整中间体的结构,最终得到产物的结构。
Swiss-Model Design
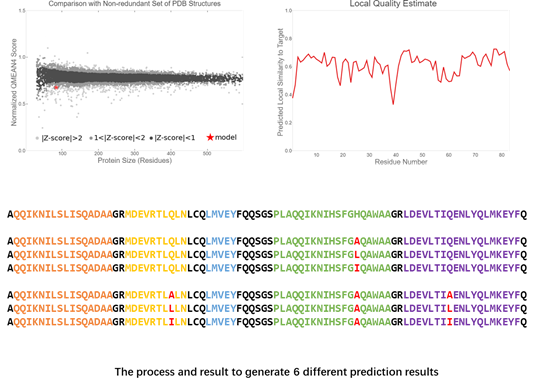
通过突变几个关键氨基酸残基,我们实现了对蛋白质口袋附近环境的修改,让中间体中吲哚环的旋转空间更大,达到了4+4反应发生的前提条件。
建模后的氨基酸网络如下图所示:
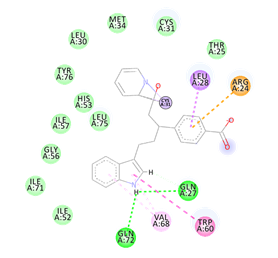
我们将这个分子进行了opt方法的高斯模拟,得到了4+4反应的八元环产物。但是因为蛋白质构象或结构优化等等问题,导致高斯结果存在不收敛的问题,这些问题将会在以后补充MD模拟与分析后加以解决。
Conclusion and Discussion
经过这次探索,我们在尝试与探索的过程中有所收获,也希望我们的成果能够给以后的研究带来帮助。首先,我们的4+4反应发现于偶然,但是经过理论上反应机理的探讨,我们发现其发生的前提条件均能够满足。而且,4+4反应尽管在自然界中很少出现,但是分步发生的4+4反应却改变了之前协同反应反Hückel芳香性质的问题。用理论的推导来指导实验或模拟的进行,能够对项目的方向把握上起到很好的作用。
其次,这次我们此次选用的Swiss-Model算得上是最为简单、但高效的同源建模方法。但为了探索更多的能够修改的范围,利用同源建模来预测目标蛋白的折叠过程能够有更多的技术来引入。从Rosetta到I-TASSER,这些用于蛋白质结构预测的方法都能够帮助我们提供更可能的酶突变结构。但是这些工具的使用都是由目标想得到的反应所决定的。所以从目标出发,先探讨反应物能够发生的不同反应,以及探索影响反应选择性的关键结构,再从关键结构出发利用bottom-up的方法进行研究,才是我们这次课题的核心思想。利用化学理论来预测可能发生的反应,再利用潜在反应的关键中间体来优化酶结构,最后得到具有全新功能的酶,这就是我们本次课题的核心思想。这样的核心思想我们希望能够用于更多的设计的方案,能够利用更多的算法作为辅助,最终能够实现酶设计的自动化、高通量化。